美国贝勒医学院章冰团队和Valentina Hoyos团队、复旦大学高强团队以及中国科学院分子细胞科学卓越创新中心高大明团队合作,通过整合10种癌症类型1043名患者的蛋白基因组学数据、细胞系的基因依赖性数据以及肿瘤抗原预测,系统地识别并实验验证了新的癌症治疗靶点。
北京时间2024年6月24日,这项研究发表在《细胞》杂志上,绘制了用于药物再利用和新疗法开发的蛋白和肽段靶点的全景图。
研究团队还在LinkedOmics工具系列 (https://www.linkedomics.org/)下创建了一个新的网络门户(https://targets.linkedomics.org/),供研究人员访问本研究中发现的蛋白和肽段靶点及其支持数据。

癌症的基因组特征研究虽然显著提升了我们对癌症生物学和基因驱动因素的理解,但目前大多数癌症患者仍然接受非特异性化疗和放疗,这些疗法的效果低且毒性高。分子靶向疗法,如小分子药物、单克隆抗体、抗体药物偶联物,蛋白质降解靶向嵌合体, 癌症疫苗和T细胞疗法,可以提高治疗效果和减少毒性。由于蛋白质是这些疗法的主要靶点,近期涌现的大规模的CPTAC蛋白基因组数据为寻找新的治疗靶点提供了重要机遇。
研究亮点
1.靶向蛋白的系统分类和定量:
研究人员系统收集了2,863个潜在的小分子靶点蛋白和膜蛋白,并将其分类为五个等级,以便于患者选择、药物再利用和新疗法开发等不同应用。
在CPTAC蛋白质组学数据中,超过80%的这些蛋白得到了量化。其中超过半数的蛋白在一种或多种癌症类型中显示出较差的mRNA-蛋白质相关性,揭示了用蛋白质组学量化药物靶点的必要性。
2.过表达和过度激活的蛋白:
研究人员分析了各种癌症类型的蛋白质组数据,以识别在肿瘤组织中相对于正常组织显著过表达且在相同癌症谱系的细胞系中有显著依赖性的蛋白。在这些蛋白中,研究团队发现了457个潜在靶点蛋白,其中有51个在至少5种癌症类型中共享,是药物再利用和新疗法开发的最佳候选者。除了蛋白丰度之外,他们的分析还结合了磷酸蛋白组数据推断的蛋白活性,揭示了通过蛋白丰度无法识别的其他药物靶点。研究人员进一步利用公共数据和新的体外和体内实验验证了这些靶点的有效性。
3.合成致死策略:
针对难以靶向的肿瘤抑制基因丢失,研究人员整合了CPTAC蛋白丰度、磷酸化位点和激酶活性数据以及DepMap基因依赖性数据,寻找与肿瘤抑制基因丧失相关的蛋白过表达,过度激活,以及升高的基因依赖性作为潜在靶点。这些靶点的抑制可能与肿瘤抑制基因的丢失形成合成致死关系。以TP53为例,研究人员发现TP53丢失突变可作为选择乳腺癌患者进行CHEK1抑制以及选择子宫内膜癌患者使用阿霉素治疗的候选生物标志物。阿霉素是一种用于子宫内膜癌的二线治疗药物,在未筛选患者中的总体反应率仅为16%。使用TP53丢失突变作为标志物筛选患者有可能大幅度提高反应率。
4.免疫疗法靶点的优先排序:
研究人员还开发了计算流程来筛选新抗原和肿瘤相关抗原作为免疫治疗的靶点。大多数预测的新抗原只存在于单个患者,但五个KRAS突变肽被预测在来自四种癌症类型的44名患者中产生新抗原,具有作为“公共”新抗原的潜在用途。研究人员还找到了140种在正常组织中表达高度局限但在CPTAC肿瘤中异常表达的蛋白。他们对七个蛋白预测了与最常见的HLA-A02等位型具有高结合亲和力的肽段并在美国和中国人群进行了实验分析。实验结果鉴定出21个来自五种蛋白的肽段,这些肽段既具有强HLA结合亲和力又具有免疫原性,可进一步作为免疫治疗靶点进行研究。
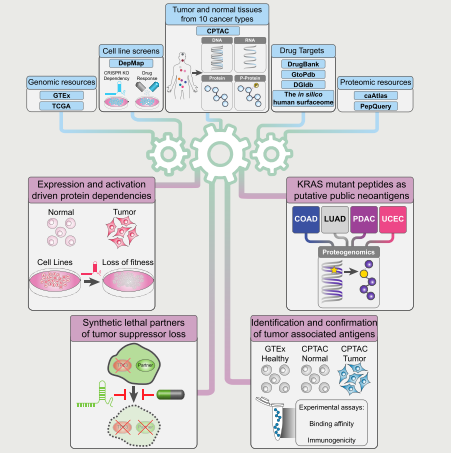
图文摘要
综上所述,该研究为癌症治疗创建了一个蛋白和肽段靶点的全景图,为未来的伴随诊断、药物再利用和新疗法的开发铺平了道路。这一开创性的研究结果展示了蛋白质组学在癌症治疗靶点识别中的巨大潜力,并为个性化医疗带来了新的希望。
来源:科学网
作者:小柯生命